记录一下使用joinmap定位目标性状位置的操作。
对目标性状进行定位使用的软件是Joinmap 4.0,网上面有很多特别版,都是可以使用的。软件界面如下
目前市面上大部人使用的应当都还是韩国忠南大学的这个授权码
做定位之前需要准备一下资料
1,目标群体相关性状的田间调查结果,群体大小建议大于200.
2,目标区段的Indel标记的电泳结果
下面开始分析
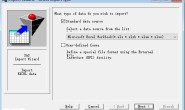
1,首先建立工程
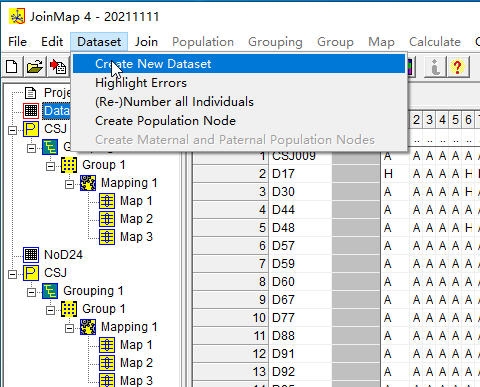
2,然后建立数据集
3,数据集的设置比较重要
Pop.name:这个可以随意设置,自己能记住就行
Pop.type:群体的类型,是F2,还是BC1等等,按照具体情况设置,我的是F2临时群体
Nr.of.loci: 这里是标记的数量,加上目标性状。如果跑了22个Indel标记,加上目标性状,那么这里就是23
Nr.of indiv.: 看名字就知道了,这里是指群体的大小,一个不能多一个不能少。我这里就是286个单株的F2群体。
Locus:分子标记的名称
Classification:空着就行,不用管他。
4,分子标记的录入
差异标记的读取:把条带与携带目标形状的亲本一致的设置为A,另一个亲本为B,杂合的为H。
目标基因的读取:与带目标形状亲本一致的为A,其余的全部为C。
这里的条带只关注与亲本相同位置的,其它位置的不用考虑。
示例如下:
有个小诀窍:
建议在excel上面录入好了之后,一次性COPY到joinmap就行了。joinmap本身的这个录入太扯了,不是很好用。
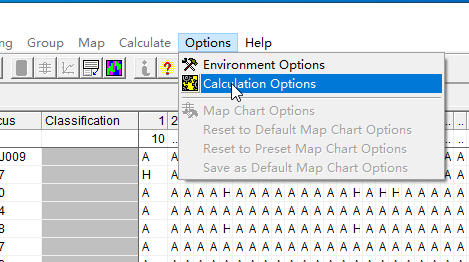
5,开始分析,首先设置运算法则
点击这个“calculation option”
定位算法(Mapping algorithm)建议选择回归算法(Regression mapping),ML mapping为最大似然算法,我测试了一下,感觉不是很行。
定位函数(Mapping function),我选择的是Kosamb’s。据悉:kosambi考虑了双交换,Haldane不考虑双交换
然后点击“ok”就行了
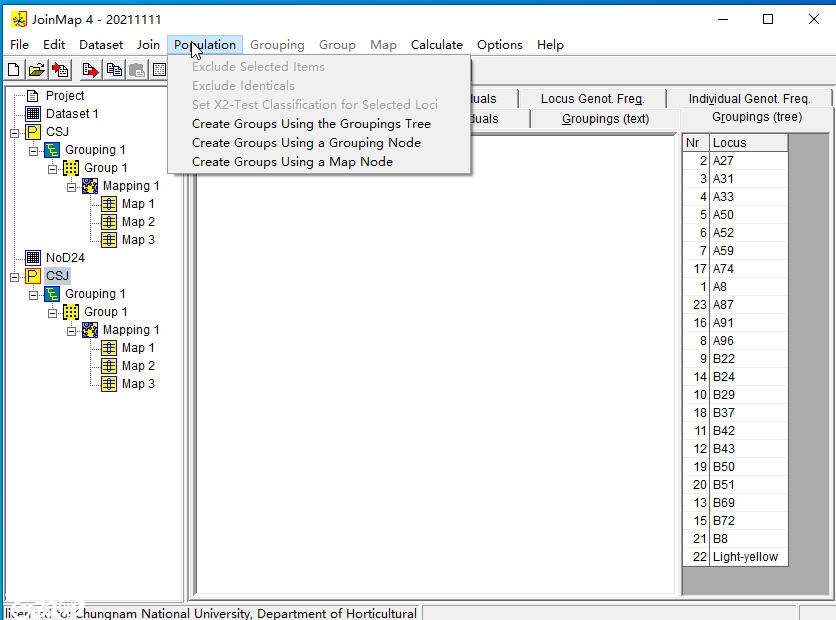
6,建立群体节点(population node)
在dataset中选择“create population Node”极可。
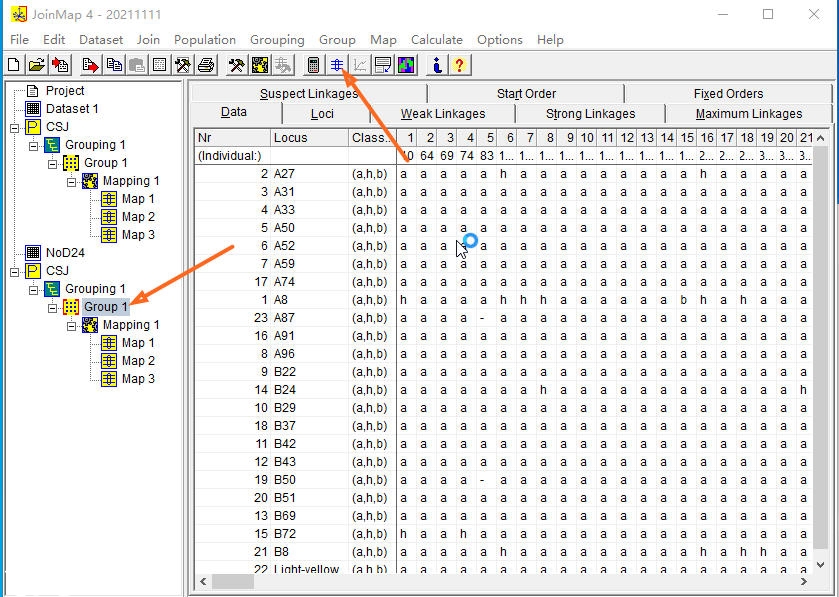
在右边框,选择Grouping(tree),然后点击上面的运算logo(类似计算器小图标)。
选择节点,使之背景变红。怎样使之变红,这里卖个小关子了。
然后选择菜单“population”,在下面选择“create groups using the grouping tree”,就会创建我们需要的分组了
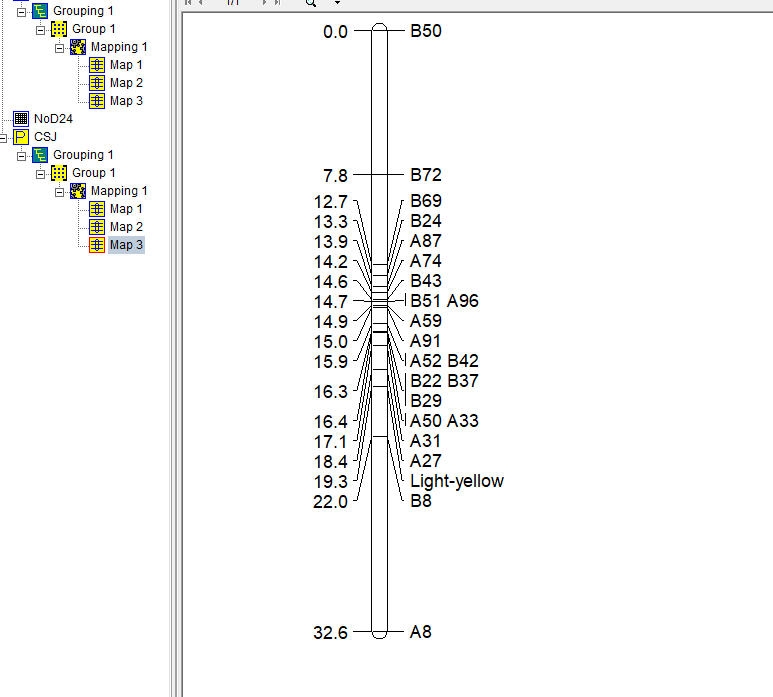
先选择group 1,然后点击上面的“Calculate map”就会构建我们的连锁图了。
大功告成额
转载请注明:百蔬君 » 【原创文章】利用joinmap 4定位连锁性状