作者:杨春文,林清 ,吴红,蒋晓英,黄任中,黄启中,吴晓莲,雷开荣
来源:中国农学通报2014,30(16):71-78
摘要:分子标记是生物学研究的一个重要工具,在作物新品种选育等方面发挥着越来越重要的作用。为了构建辣椒分子标记辅助育种技术体系,以实现常规育种技术与现代生物技术的有机结合,归纳了分子标记技术在辣椒种质遗传多样性、分子连锁图谱构建、分子标记辅助选择育种和基因定位等方面的已有研究成果;认为分子标记技术在辣椒上的应用研究系统性还不强、基础分子信息比较缺乏、遗传图谱还不精细,建议在实用新型分子标记开发、核心种质分子信息库建立、优良作图群体及精细遗传图谱构建等方面加强系统深入的研究。
0 引言
辣椒(Capsicum spp.),又名番椒、海椒、辣子、辣茄等,属于茄科(Solanaceae)辣椒属(Capsicum L.),是一种重要的蔬菜作物和调味品,广泛栽培于南北美、欧洲、亚洲和大洋洲等世界各地。辣椒的营养价值很高,维生素C含量在蔬菜中占首位,是番茄的7~15倍;辣椒素是主要的辣味来源,有药理作用和工业用途[1]。中国是世界上辣椒栽培面积最大的国家,年播种面积130 万hm2~160 万hm2,总产值达270 亿~300亿元[2];随着社会经济的不断发展,人们对辣椒的需求呈现多样化的发展趋势,虽然在辣椒的遗传改良方面已经取得了很大进展,但由于可直接利用的种质资源有限及常规育种技术的局限,一定程度上限制了辣椒新品种培育的进程,新品种在品质、产量、抗性等方面表现徘徊,鲜有重大突破。
DNA 分子标记是生物学研究的一个重要工具,自Grodzicker[3]创立RFLP技术以来,已经有多种DNA分子标记如限制性片段长度多态性(RFLP)、随机扩增多态性(RAPD),简单串联重复序列(SSR)和扩增片段长度多态性(AFLP)等被发明和应用,促进了作物遗传改良研究。在辣椒分子标记研究方面,自1993 年美国康乃尔大学的Prince 等[4]利用RFLP 和同工酶技术构建了世界上第一个辣椒的分子遗传图谱以来,分子标记技术在辣椒种质遗传多样性[5-17]、遗传图谱构建[25-38]、分子标记辅助选择[39-44]等方面取得了长足进展(表1),为辣椒遗传改良提供了强大的技术支撑。但是,与水稻、玉米等粮食作物以及甘蓝、黄瓜等蔬菜作物比较,辣椒的分子标记应用研究还存在不系统、不深入的问题,主要表现在实用新型标记开发力度较弱、核心种质及骨干亲本的分子信息缺乏、优良作图群体少、遗传图谱还不精细等。为进一步提升辣椒的遗传改良水平,拟借助现代生物技术手段,构建实用的辣椒分子标记辅助育种技术体系,本研究系统回顾了近年来国内外辣椒分子标记研究的主要研究进展,为开展相关工作积累了丰富的技术资料并奠定了相关的理论基础。

1 辣椒种质遗传多样性的分子标记分析
作物种内的遗传多样性或称遗传变异,是作物进化的基本前提,同时也是遗传改良及新品种培育的基础。研究遗传多样性的方法随着生物学尤其是遗传学和分子生物学的发展而不断创新和完善,已从形态学水平、细胞学(染色体)水平、生理生化(酶)水平逐渐发展到了分子水平。
目前,分子标记技术已经被广泛的运用到辣椒种质资源遗传多样性的研究中。Tam 等[5]应用SSAP、AFLP和SSR分子标记技术研究了番茄和辣椒的遗传关系;周晶等[6]利用SSR 标记分析了89 份辣椒种质的遗传多样性,在87 对能扩增出条带的SSR 引物中,有52 对引物具有多态性,经SSR 分子标记聚类分析,在遗传相似系数为0.60 时,可以将栽培种和野生种区分开来,在遗传相似系数为0.80 时,能将不同果实类型的种质分别聚类;罗玉娣等[7]利用21 对SSR 引物,将33个辣椒材料分为5 类,同一种内的辣椒种质资源基本聚在一起;杜晓华等[8]利用RSAP和SSR技术对辣椒优良自交系间遗传距离进行评估并比较两者的差异,认为RSAP标记比SSR具有更高的位点和多态性检测能力,但是基于RSAP 的遗传距离和基于SSR 的遗传距离相关性较高,2 种标记的聚类结果基本一致。Paran等[9]利用RAPD 和AFLP 技术鉴定辣椒资源的遗传关系;出于品种保护的需要,Lefebvre 等[10]利用AFLP、RAPD 和表型特征对47 份辣椒近交系的遗传距离进行测定,并将这些近交系划分为5 个类群;Lanteri 等[11]也利用RAPD和AFLP技术对意大利西北部当地辣椒’Cuneo’的5 个变异种进行了遗传分析,认为41.6%的可能性是种间遗传多样性,58.4%是种内遗传多样性;Geleta 等[12]则利用表型性状和AFLP 标记研究了杂种优势和遗传距离之间的关系,有效的将杂合子区分开。在国内,马艳青等[13]和王玲等[14]利用RAPD 技术对辣椒种质资源进行了多样性分析,李晴等[15] 用RAPD技术,将37 份辣椒材料分为5 个类群,每个类群内的种质材料间亲缘关系较近。
2 辣椒品种真实性及种子纯度的分子标记鉴定
品种真实性及种子纯度是保证辣椒果实产量和质量的重要参数。随着分子生物学技术的飞速发展,分子标记技术已成为作物纯度鉴定的重要手段,并在玉米、小麦、水稻、大豆、棉花、西瓜、甜瓜等作物的种子质量监控方面得到广泛应用;在辣椒的品种鉴定中,也正在逐渐建立和完善相关的技术体系。宋小丽等[16]利用AFLP 分子标记对12 个亲缘关系密切、形态相近的辣椒品种进行鉴定,并成功利用不同的引物来区分鉴定;Livneh 等[18]利用RFLP 标记鉴定杂交辣椒和其父母本之间的区别,并成功的将其改造成一个简单适用的PCR 检测方法;Ballester 等[19]利用RAPD 和SPARS 技术探索种子纯度鉴定的方法,筛选到一条能够准确区分5 个F1杂交种及相应父母本的特征引物;Ilbi[20]利用RAPD技术研究了辣椒变异性和种子纯度,并成功筛选到品种特异性引物和能区分所有参试杂交种的引物;李亚利等[21]用SRAP分子标记研究了2 个辣椒品种F1的纯度,结果与田间试验非常接近;白占兵等[22]用116 对多态性较高的引物对12 个辣椒推广品种及其父本、母本进行了PCR差异性研究,筛选出6 对能够区分每个F1品种及其父母本的引物;他们还利用SSR标记对32 个杂交种及其父母本进行鉴定,筛选出5 对特征引物,能够区别参试杂交种及其父本、母本,初步建立了基于SSR分子标记技术的辣椒种子纯度鉴定体系,为辣椒种子纯度鉴定工作提供了技术支撑[23]。
3 辣椒分子遗传图谱的构建
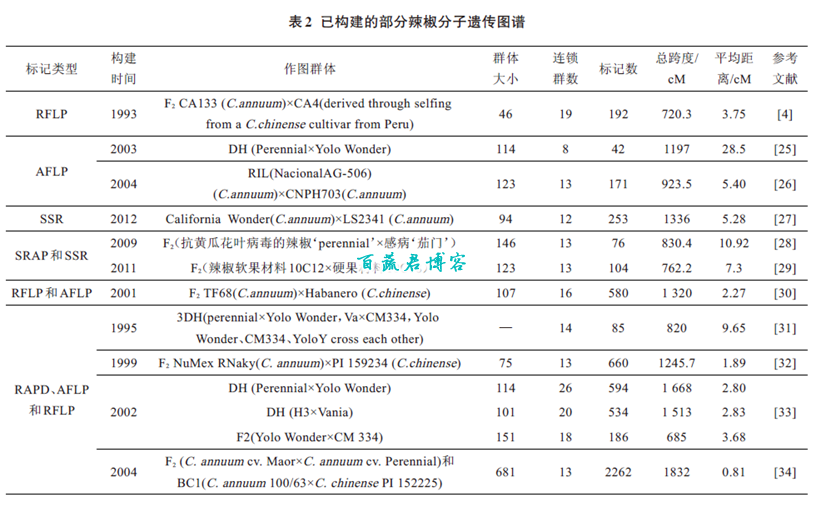
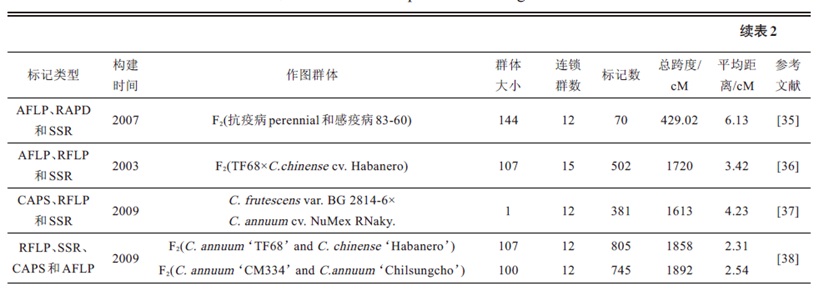
遗传图谱的构建是基因组研究中的重要环节,是基因定位与克隆乃至基因组结构及功能研究的基础。1984 年,Tanksley等[24]根据形态同工酶标记等特征,构建了第1 张辣椒遗传连锁图谱,该图谱明确了分布在4个连锁群上的14 个同工酶的位点,为辣椒基因组的研究奠定了基础。随后,分子标记技术被广泛的应用到辣椒的遗传连锁图谱构建中。最初是用单一分子标记进行连锁图的构建,如Prince 等[4]在1993 年应用RFLP构建了第1 张辣椒分子标记连锁图谱,Wang[26]利用AFLP 技术构建辣椒连锁图谱,而Mimura 等[27]首次建立了比较完整的SSR分子标记连锁图,该连锁图谱包含253 个分子标记(151 SSRs),覆盖长度为1336 cM,包含了12 个连锁群(表2)。鉴于单分子标记在连锁图谱构建方面还存在一些缺陷,如标记数相对较少、总跨度较小、平均距离大,不利于高精密度连锁图谱的构建,混合分子标记连锁图谱的构建得以发展。刘军等[29]以辣椒软果材料10C12 和硬果材料10C08 杂交获得的F2分离群体为作图群体,利用SRAP 与SSR 分子标记构建了一张包含26 个SSR 标记和78 个SRAP 标记的辣椒分子遗传连锁图谱;而安静等[35]则以抗疫病材料perennial 和感疫病材料杂交得到的F2群体为试材,利用AFLP、RAPD、SSR 分子标记,采用JoinMap3.0 构建辣椒分子连锁遗传图谱,该图谱包括12 个连锁群、70个标记位点;Lee 等[38]利用RFLP、SSR、CAPS、AFLP等分子标记技术建立一个比较完整的辣椒分子遗传连锁图谱,该连锁图谱包含了12 个连锁群,共1962 个分子标记、覆盖长度为1858 cM,平均遗传距离2.31 cM。表2 为近年来已构建的辣椒分子遗传图谱的基本情况。
4 辣椒相关性状的基因定位及分子标记辅助育种
4.1 辣椒抗病性相关基因定位与分子标记选择
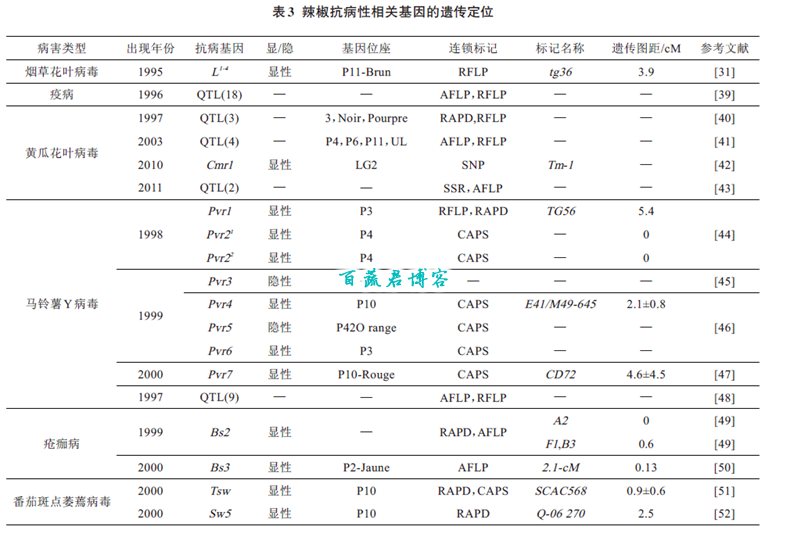
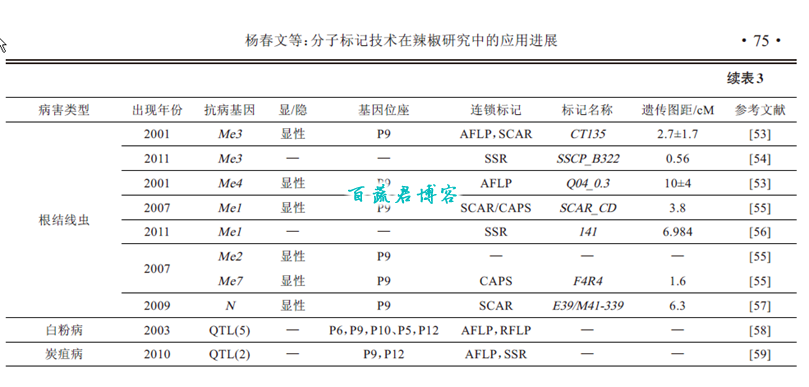
辣椒的主要病害有病毒病、疫病、疮痂病、白粉病等,它们的发生程度直接影响辣椒果实的产量和品质。利用现代生物技术手段,发掘、定位抗病性相关基因,实现分子标记辅助育种,对促进辣椒种质创新及抗病新品种培育具有重要作用。Kang 等[42]在辣椒抗病株系’Bukang’中发掘出一个显性基因Cmr1,并将其定位于着丝粒的LG2区,它对CMVKorean和CMVFNY 2 种菌株抗性显著;赵娟等[43]将抗黄瓜花叶病毒的基因QTL 定位在第1、4、7 连锁群上,抗性贡献率分别为12.7%、38.8%、11.0%。在辣椒抗根结线虫病基因的方面,对已经命名的6 个显性单基因N、Me1、Me2、Me3、Me4、Me7 进行了分子标记定位[53-57]。表3 为已经定位的辣椒烟草花叶病等9 种病害相关基因的基本信息。
4.2 辣椒育性及果实性状相关基因定位与分子标记选择
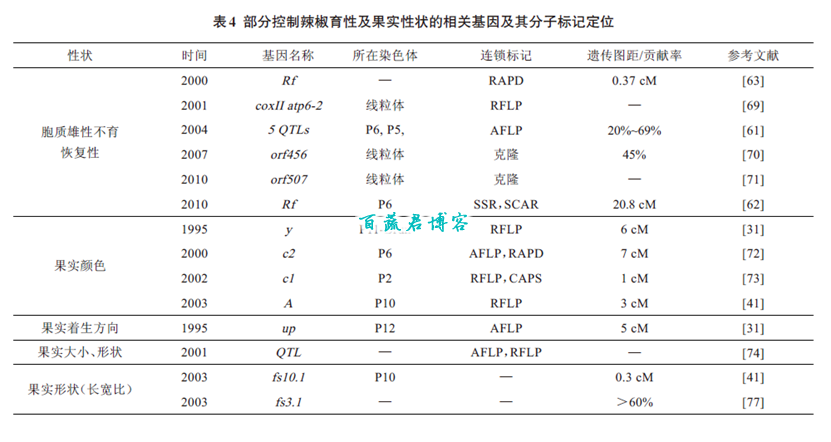
辣椒雄性不育有2 种,一种是细胞核雄性不育(genic male sterility,GMS),由单隐性基因控制;另一种是细胞质雄性不育(cytoplasmic male sterility,CMS)。细胞质雄性不育广泛存在于高等植物中,其调控机理相对复杂,有单基因和双基因的争论,普遍认为是由单个主效基因控制、多个微效基因修饰作用的结果[60]。王立浩等[61]主要利用DH分离群体,通过分析育性的分离情况,评价了CMS 的遗传规律,得出恢复性由主效基因和4 个微效基因控制的结论,并将主效基因定位到P6 染色体上;而杨娟等[62]以辣椒不育系113A、恢复系139 及其F2代群体为材料,对辣椒恢复基因进行SSR 标记定位,结果发现引物AF208834 和Rf 基因连锁,遗传距离为20.8 cM,笔者同时将SSR标记和SCAR标记结合起来,建立了与育性基因有关的多重PCR技术,显著提高了辣椒雄性不育三系的选择效率;张宝玺等[63]利用RAPD技术发现2 个与恢复基因紧密连锁的标记OP131400和OW19800,他们分别位于Rf 基因的两侧,遗传距离为0.37 和8.12 cM,2005 年Kim[64]将RAPD 标记OPP131400 转化为OPP13-CAPS(HinfI)标记。Lee 等[65]和Gulyas 等[66]将RAPD 标记OPT-02/570 转换成一个同Rf 基因紧密连锁的STS 标记(CRF-SCAR),此标记距离Rf 基因5 cM;Kim 等[67]发现一个遗传距离更近的显性分子标记AFRF8-CAPS (1.8 cM),此标记可用于F1杂交种子产量和育种工作的检测;Lee 等[68]发掘了与pr 基因相连锁的分子标记PR-CAPS。此外,还利用分子标记技术定位了辣椒果实相关性状基因,如果实颜色[31,41,72- 73]、果实大小与形状[41,70,77]、辣味[76,78]、辣椒红素[79]等(见表4)。
5 展望
随着分子生物学的不断发展,分子标记技术在支撑作物遗传改良方面将发挥越来越重要的作用。虽然,分子标记技术在辣椒遗传多样性分析、种子质量鉴定、遗传图谱构建、基因精细定位和分子标记辅助育种等方面取得了一些进展,但是,与其他作物比较,辣椒的分子标记研究及应用还有较大差距。借鉴水稻、玉米等作物研究方面的经验,还需重点推进以下工作:
一是加强实用新型标记的开发。目前辣椒遗传多样性的标记多为RAPD、AFLP 等标记,标记的种类比较单一,操作复杂,一些更稳定的、易于操作的标记如SCAR、RSAP、CAPS等还有待于开发研究。
二是加强辣椒种质特别是核心种质及骨干亲本的分子信息库建立,有利于分子标记辅助选择技术体系的构建,并进而更好实现与常规育种的有机结合。
三是加强多种类型作图群体的构建。已构建的辣椒分子遗传图谱仍比较粗糙,遗传距离跨度小,平均遗传距离大,所用群体多为F2代等临时群体,这不利于精细图的构建;下一步,结合常规育种,要充分利用辣椒的种质资源,构建近等基因系、单片段代换系等永久群体,用于构建包含分子标记种类更加丰富、数量更加庞大的辣椒遗传图谱,以满足分子辅助育种的需求。
参考文献
[1] 方宣钧,吴为人,唐纪良.作物DNA标记辅助育种[M].北京:科学出版社,2001:7-21.
[2] 李雪峰,王文英,白占兵,等. 3 种内包装对辣椒种子发芽的影响[J].中国农学通报,2009,25(18):311-313.
[3] Grodzicker T, Williams J, Sharp P, et al. Physical mapping of temperature- sensitive mutations of adenoviruses[J].Cold Spring Harbor Symposia on Quantitative Biology, 1974,39:439-446.
[4] Prince J P, Pochard E, Tanksley S D. Construction of a molecular linkage map of pepper and a comparision of synteny with tomato[J].Genome, 1993, 36: 404-417.
[5] Tam S M, Mhiri C, Vogelaar A, et al. Comparative analyses of genetic diversities within tomato and pepper collections detected by retrotransposon- based SSAP, AFLP and SSR[J].The oretical and Applied Genetics, 2005, 110(5): 819-831.
[6] 周晶,沈火林,杨文才,等.辣椒遗传多样性的SSR分析[J].华北农学报,2009,24: 62-67.
[7] 罗玉娣,李建国,李明芳.用SSR 标记分析辣椒属种质资源的遗传多样性[J].生物技术通报,2006:337-341.
[8] 杜晓华,王得元,巩振辉.基于RSAP和SSR的辣椒优良自交系间遗传距离的估计与比较[J].西北农林科技大学学报,2007,35:97-102.
[9] Paran I, Aftergoot E, Shifriss C. Variation in Capsicum annuum revealed by RAPD and AFLP marker[J].Euphytica, 1998, 99(3):167-173.
[10] Lefebvre V, Goffinet B, Chauvet J C, et al. Evaluation of genetic distances between pepper inbred lines for cultivar protection purposes: comparison of AFLP, RAPD and phenotypic data[J]. Theoretical and Applied Genetics, 2001,102(5):741-750.
[11] Lanteri S, Acquadro A, Quagliotti L, et al. RAPD and AFLP assessment of genetic variation in a landrace of pepper (Capsicum annuum L.), grown in North-West Italy[J].Genetic Resources and Crop Evolution, 2003,50(7):723-735.
[12] Geleta L F, Labuschagne M T, Viljoen C D. Relationship between heterosis and genetic distance based on morphological traits and AFLP markers in pepper[J].Plant Breeding, 2004,123(5): 467-473.
[13] 马艳青,刘志敏,邹学校.辣椒种质资源的RAPD分析[J].湖南农业大学学报,2003,29: 120-123.
[14] 王玲,郭金贵,焕钟雄,等.辣椒遗传多样性的RAPD分析[J].福建农林大学学报,2003,32(2):213-216.
[15] 李晴,张学时,张广臣,等.辣椒种质遗传多样性的RAPD分析[J].北方园艺,2010,22: 118-122.
[16] 宋小丽,柴伟国,董德坤,等.12 个亲缘关系密切辣椒品种的AFLP鉴定[J].浙江农业科学,2008,3:259-262.
[17] Lefebvre V, Palloix A, Rives M. Nuclear RFLP between pepper cultivars (Capsicum annuum L.)[J].Euphytica, 1993,71(3):189-199. [18] Livneh O, Vardi E, StramY, et al. The conversion of a RFLP assay into PCR for the determination of purity in a hybrid pepper cultivar [J].Euphytica, 1992, 62(2): 97-102.
[19] Ballester J, Vicente M C. Determination of F1 hybrid seed purity in pepper using PCR- based markers[J].Euphytica, 1998, 103(2):223-226.
[20] Ilbi H. RAPD markers assisted varietal identification and genetic purity test in pepper, Capsicum annuum[J].Scientia Horticulturae,2003,97:211-218.
[21] 李亚利,扈新民,赵丹,等.运用SRAP 分子标记鉴定辣椒杂交种纯度[J].中国农学通报,2010,26(24): 67-70.
[22] 白占兵,李雪峰,戴雄泽.利用SSR 分子标记建立辣椒纯度鉴定体系[J].辣椒杂志,2010,1: 32-34.
[23] 白占兵,李雪峰,谭新跃,等.辣椒杂交种纯度快速鉴定体系建立与应用分析[J].中国农学通报,2012,28(16):277-281.
[24] Tanksley S D, Bernatzky R, Lapitan N L, et al. Construction of a melecular linkage map of pepper and tomato[J].Proceedings of the National Academy of Sciences, USA,1988,36:401-417.
[25] 张宝玺,王立浩,黄三文,等.辣椒分子遗传图谱的构建和胞质雄性不育恢复性的QTL定位[J].中国农业科学,2003,36(7):818-822.
[26] Wang Y Q. Construction of a molecular linkage map of pepper (Capsicum spp.) using AFLP markers and RIL population,[EB/OL] 2004, http://www.arc-acrdc.org/html-files/molecular. html.
[27] Mimura Y, Inoue T, Minamiyama Y,et al. An SSR- based genetic map of pepper (Capsicum annuum L.) serves as an anchor for the alignment of major pepper maps[J].Breeding Science, 2012, 62: 93- 98.
[28] 赵娟. 辣椒分子连锁图谱的构建及抗黄瓜花叶病毒QTL定位[D].呼和浩特:内蒙古大学,2009.
[29] 刘军,张维,沈火林.辣椒遗传连锁图谱的构建及果实硬度的QTL分析[J].中国蔬菜,2011(8):17-21.
[30] Kang B C, Nahm S H, Huh J H, et al. An interspecific (Capsicum annuum × C. chinense) F2 linkage map in pepper using RFLP and AFLP markers[J].Theoretical and Applied Genetics, 2001,102:531-539.
[31] Lefebvre V, Palloix A, Caranta C, et al. Construction of an intraspecific integrated linkage map of pepper using molecular markers and doubled- haploid progenies[J].Genome, 1995,38:112-121.
[32] Livingstone K D, Lackney V K, Blauth J R, et al. Genome mapping in Capsicum and the evolution of genome structure in Solanaceae[J]. Genetics, 1999,152:1183-1202.
[33] Lefebvre V, Pfieger S, Thabuis A, et al. Towords the saturation of the pepper linkage map by alignment of three intraspecific maps including known-function genes[J].Genome, 2002,45:839-854.
[34] Paran I, Voort JR, Lefebvre V, et al. An integrated genetic linkagemap of pepper (Capsicum spp.) [J].Molecular Breeding, 2004,13:251-261.
[35] 安静,胡勇胜,张宝玺,等.辣椒分子连锁遗传图谱的构建及抗疫病QTL定位[J].中国蔬菜,2007,10: 9-12.
[36] Lee J M, Nahm S H, Kim Y M, et al. Characterization and molecular genetic mapping of microsatellite loci in pepper[J]. Theoretical and Applied Genetics, 2004,108:619-627.
[37] Wu F N, Eannetta N T, Xu Y M, et al. A COSII genetic map of the pepper genome provides a detailed picture of synteny with tomato and new insights into recent chromosome evolution in the genus Capsicum[J].Theoretical and Applied Genetics, 2009,118(7):1279-1293.
[38] Lee H R, Bae I H , Park S W, et al. Construction of an integrated Pepper map using RFLP, SSR, CAPS, AFLP, WRKY, rRAMP, and BAC end sequences[J].Moleculars and Cells, 2009,27:21-37.
[39] Lefebvre V, Palloix A. Both epistatic and additive effects of QTLs are involved in polygenic induced resistance to disease: a case study the interaction pepper- phytophthora capsici Leonian[J].Theoretical and Applied Genetics, 1996,93:503-511.
[40] Caranta C, Palloix A, Lefebvre V, et al. QTLs for a component of partial resistance to cucumber mosaic virus in pepper: restriction of virus installation in host- cells[J].Theoretical and Applied Genetics, 1997,94:431-438.
[41] Chaim A B, Borovsky Y, Jong W D, et al. Linkage of the A locus for the presence of anthocyanin and fs10.1 a major fruit-shape QTL in pepper[J].Theoretical and Applied Genetics, 2003,106:889-894.
[42] Kang W H, Hoang N H, Yang H B, et al. Molecular mapping and characterization of a single dominant gene controlling CMV resistance in peppers (Capsicum annuum L.) [J].Theoretical and Applied Genetics, 2010, 120(8):1587-1596.
[43] 赵娟,王立浩,毛胜利,等. 辣椒抗黄瓜花叶病毒QTL分析[J].中国蔬菜,2011,1(18):30-35.
[44] Murphy J F, Blauth J R, Livingstone K D, et al. Genetic mapping of the pvr1locus in Capsicum spp. And evidence that distinct potyvirus resistance loci control responses that differ at the whole plant and cellular level[J].Molecular Plant-Microbe Interactions, 1998,11(10):943-951.
[45] Caranta C, Nemouchi G, Daubèze A M, et al. Resistance to PepMoV and PVY- 0 from Avelar are controlled by distinct recessive genes and evidence for independence between pvr3 and pvr5[J].Capsicum and Eggplant Newslett, 1999,19:63-65.
[46] Caranta C, Thabuis A, Palloix A. Development of a CAPS marker for the Pvr4 locus: a tool for resistance to pyramiding potyvirus resistance genes in pepper[J].Genome, 1999,42:1111-1116.
[47] Grube R C, Blauth J R, Arnedo M S, et al. Identification and comparative mapping of a dominant potyvirus resistance genecluster in Capsicum[J].Theoretical and Applied Genetics, 2000,101:852-859.
[48] Caranta C, Lefebvre V, Palloix A. Polygenic resistance of Pepper to Potyviruses consists of a combination of isolate-specific and broadspectrum quantitative trait loci[J].Molecular Plant- Microbe Interactions, 1997,10(7):872-878.
[49] Tai T, Dahlbeck D, Stall R E, et al. High- resolution genetic and physical mapping of the region containing the Bs2 resistance gene of pepper[J].Theoretical and Applied Genetics, 1999, 99:1201-1206.
[50] Pierre M, Noël L, Lahaye T, et al. High-resolution genetic mapping of the pepper resistance locus Bs3 governing recognition of the Xanthomonas campestris pv vesicatora Avr Bs3 protein[J].Theoretical and Applied Genetics, 2000,101:255-263.
[51] Moury B, Pflieger S, Blattes A, et al. A CAPS marker to assist selection of tomato spotted wilt virus (TSWV) resistance in pepper [J].Genome, 2000, 43:137-142.
[52] Jahn M, Paran I, Hoffmann K, et al. Genetic mapping of the Tsw for resistance to the tospovirus tomato spotted wilt virus in Capsicum spp. and its relationship to the Sw-5 gene for resistance to the same pathogen in tomato[J].Molecular Plant-Microbe Interactions, 2000,13(6): 673-682.
[53] Djian-Caporalino C, Pijarowski L, Fazari A, et al. High-resolution genetic mapping of the pepper (Capsicum annuum L.) resistance loci Me3 and Me4 conferring heat- stable resistance to root- knot nematodes (Meloido- gyne spp.) [J].Theoretical and Applied Genetics, 2001,103:592-600.
[54] 许小艳,刘峰,康厚祥,等.辣椒抗根结线虫基因Me3的精细定位[J].园艺学报,2011,38(2):288-294.
[55] Djian- Caporalino C, Fazari A, Arguel M J, et al. Root- knot nematode (Meloidogyne spp.) Me resistance genes in pepper (Capsicum annuum L.) are clustered on the P9 chromosome[J].Theoretical and Applied Genetics, 2007, 114: 473-486.
[56] 张宇,张晓芬,陈斌,等.与辣椒抗根结线虫基因Me1 紧密连锁的EST-SSR标记开发[J].核农学报,2011,25(5): 933-938.
[57] Wang L H, Gu X H, Hua M Y, et al. A SCAR marker linked to the N gene for resistance to root knot nematodes (Meloidogyne spp.) in pepper (Capsicum annuum L.) [J].Scientia Horticultrae, 2009, 122:318-322.
[58] Lefebvre V, Daubèze A M, Voort J R, et al. QTLs for resistance to powdery mildew in pepper under natural and artificial infections[J].Theoretical and Applied Genetics, 2003, 107: 661-666.
[59] Lee J, Hong J H, Do J W, et al. Identification of QTLs for resistance to Anthracnose to two Colletotrichum species in pepper[J].Journal of Crop Science and Biotechnology,2010,13(4):227-233.
[60] 王立浩,张宝玺,杜永臣.辣椒基因遗传定位及分子遗传图谱的研究进展[J].园艺学报,2005,32:1147-1154.
[61] Wang L H, Zhang B X, Lefebvre V, et al. QTL analysis of fertility restoration in cytoplasmic male strile pepper[J].Theoretical and Applied Genetics, 2004, 109: 1058-1063.
[62] 杨娟,王雯,沈火林.辣椒恢复基因SSR 标记定位及分子标记辅助选择育种[J].中国瓜菜,2010,23(5): 1-5.
[63] Zhang B X, Huang S W, Yang G M, et al. Two RAPD markers linked to a major fertility restorer gene in pepper[J].Euphytica, 2000, 113: 155-161.
[64] Kim D S. Development of RAPD and AFLP markers linked to fertility restorer (Rf) gene in chili pepper (Capsicum annuum L.)[J]. Thesis, 2005, Seoul National University.
[65] Lee J S, Lee K H, Kim Y S, et al. Detection of individuals restoring fertility by DNA fragment converted into STS (sequence tagged site) in red pepper[J].Plant Resources, 2004,7:136-140.
[66] Gulyas G, Pakozdi K, Lee J S, et al. Analysis of fertility restoration by using cytoplasmic male- sterile red pepper (Capsicum annuum L.) lines[J].Breed Science, 2006, 56:331-334.
[67] Kim D S, Kim D H, Yoo J H, et al. Cleaved amplified polymorphic sequence and amplified fragment length polymorphism markers linked to the fertility restorer gene in chili pepper (Capsicum annuum L.)[J].Molecules and Cells, 2006, 21:135-140.
[68] Lee J, Yoon J B, Park H G. A CAPS marker associated with the partial restoration of cytoplasmic male sterility in chili pepper (Capsicum annuum L.)[J].Molecular Breeding, 2008,21:95-104.
[69] Kim D H, Kang J G, Kim S, et al. Identification of coxII and atp6 regions as associated to CMS in Capsicum annuum by using RFLPand long accurate PCR[J].J Korean Soc Hortic Sci, 2001,42:121-127.
[70] Kim D H, Kang J G, Kim B D. Isolation and characterization of thecytoplasmic male sterility- associated orf456 gene of chili pepper(Capsicum annuum L.) [J].Plant Molecular Biology, 2007,63:519-532.
[71] Gulyas G, Shin Y, Kim Hoytaek, et al. Altered transcript reveals anOrf507 sterility-related gene in Chili Pepper (Capsicum annuum L.)[J].Plant Molecular Biology Reporter, 2010,28:605-612.
[72] Thorup T A, Tanyolac B, Livingstone K D, et al. Candidate geneanalysis of organ pigmentation loci in the Solanaceae[J].Proceedings of the National Academy of Sciences, USA, 2000,97:11192-11197.
[73] Blum E, Liu K, Mazourek M, et al. Molecular mapping of the Clocus for presence of pungency in Capsicum[J].Genome,2002,45:702-705.
[74] Chaim A B, Grube R C, Lapidot M, et al. Identification ofquantitative trait loci associated with resistance to cucumber mosaicvirus in Capsicum annuum[J].Theoretical and Applied Genetics,2001,102:1213-1220.
[75] Rao G U, Paran I. Polygalacturonase: a candidate gene for the soft flesh and deciduous fruit mutation in Capsicum[J].Plant MolecularBiology, 2003,51:135-141.
[76] Blum E, Mazourek M, O’Connell M, et al. Molecular mappingof capsaicinoid biosynthesis genes and quantitative trait locianalysus for capsaicinoid content in Capsicum[J].Theoreticaland Applied Genetics, 2003,108:79-86.
[77] Chaim A B, Borovsky Y, Rao G U, et al. fs3.1: a major fruitshape QTL conserved in Capsicum[J].Genome, 2003,46:1-9.
[78] Stewart J C, Kang B C, Liu K, et al. The Pun1 gene for pungency inpepper encodes a putative acyltransferase[J]. The Plant Journal,2005,42:675-688.
[79] 张芳芳,张正海,毛胜利,等.辣椒成熟果实中辣椒红素含量的QTL 定位[J].中国蔬菜,2010(20):14-18.
转载请注明:百蔬君 » 美文分享:分子标记技术在辣椒研究中的应用进展



