王立浩 张宝玺 杜永臣
来源:园艺学报
摘 要:对近几年辣椒基因遗传定位、遗传图谱构建及遗传图谱比较研究进行了综述, 阐述了这一领域的主要研究进展, 指出目前研究中存在的主要问题, 并对今后我国辣椒分子标记辅助育种的发展方向作了展望。
辣椒(CapsicumannuumL.)在世界上广泛栽培。中国是世界上辣椒栽培面积最大的国家, 2002
年的栽培面积达到了130万hm2 , 产量约2 819万t〔1〕。重要性状的遗传定位和遗传图谱的研究对于辣椒遗传育种有重要意义。早在20世纪50 ~ 60年代, 遗传育种学家就开始利用辣椒的形态标记将部分控制果实形状和颜色的基因进行了定位〔2〕。由于形态标记数目较少, 形态标记遗传图谱在辣椒育种中的应用非常有限。20世纪80年代, 同工酶(isozymes)、限制性片段长度多态(RFLP)等分子生物技术的发展, 为构建遗传图谱提供了稳定的易于作图的生化和分子标记, 突破了传统形态多态的限制。90年代以来, 随着RAPD、SSR、AFLP等分子标记技术的发明和应用, 大大丰富了可利用的分子标记的种类, 辣椒分子标记和分子遗传图谱得到了很大的发展。由于分子标记突破了传统标记数目的限制, 而差异本身来自于DNA, 由此建立的遗传定位和图谱为解决多基因控制的优异农艺性状的定向转育提供了有效的工具。在研究辣椒分子遗传图谱的同时, 遗传图谱的比较研究也在展开。通过茄科蔬菜的图谱比较, 清楚了其进化关系, 以及基因在茄科不同作物之间的分布情况等。不久前康乃尔大学建立了茄科作物基因组学网络(http: //sgn.cornell.edu), 集中了关于茄科作物基因组学中的大量数据库, 进一步方便世界各国的学者进行比较和研究。本文就辣椒的基因定位和分子图谱的研究及其与茄科蔬菜图谱的比较作一综述。
1 辣椒重要农艺性状的分子遗传定位
1.1 抗病性及抗虫性
辣椒的病虫害较多。主要的病害有病毒病、疫病、疮痂病、白粉病等, 主要的虫害有茶黄螨、蚜虫、白粉虱、蓟马、线虫等。近年来抗病基因的遗传定位发展很快, 利用部分抗源材料, 抗病毒病、疫病、疮痂病、白粉病、及线虫等的基因相继得到了定位。
1.1.1病毒病 病毒病是辣椒上的重要病害, 世界上有45种之多。中国至今年共发现了8种主要的病毒侵染辣椒, 即黄瓜花叶病毒(CMV)、烟草花叶病毒(TMV)、马铃薯X病毒(PVX)、马铃薯Y病毒(PVY)、蚕豆萎蔫病毒(BBWV)、苜蓿花叶病毒(AMV)、烟草蚀纹病毒(TEV)、烟草脆裂病毒(TRV)。其中CMV、TMV、PVY发生较为普遍, 目前均完成了遗传定位。
烟草花叶病毒:根据与L基因的抗性情况, 将烟草花叶病毒分为了4 个小种——— P0 , P1 , P1-2 ,P1-2-3 。已知L0 基因不抗TMV的任何小种;L1 基因抗P0 小种, 但不抗P1 小种;L2 基因抗P1 小种,但不抗P1-2小种;L3 基因抗P1-2 , 但不抗P1-2-3小种;L4 基因抗P1-2-3小种〔3〕。L0 ~ 4基因的定位研究较早,20世纪80年代, Boukema等〔3〕研究发现L0 ~ 4为复等位基因, 位于基因组内的同一位点, 其显性关系为L4 >L3 >L2 >L1 >L0 。1995年, Lefebvre等把L定位到分子遗传图谱第P11 -Brun染色体上〔4〕(表1)。
黄瓜花叶病毒:国际上多数学者认为目前发现的辣椒抗CMV材料的抗性是由多个基因控制的。
1997年, Caranta等利用抗病材料Perennial和感病材料YoloWonder的DH群体进行了抗CMV的QTL分析, 得到了3个QTL, 一个在LG3上, 与多个分子标记有显著的相关关系, 为连锁的标记簇, 另一个位于染色体Noir上;再一个位于染色体Pourpre上与QTL(2)上位互作〔5〕。2001年, Chaim等利用抗病材料Perennial和感病材料Maor建立了F2 群体, 又一次进行了定位〔6〕, 结果发现了4 个QTL, 分别位于第4、6、11和UL连锁群上。其中除了位于第6染色体上的QTL很可能同Caranta等〔5〕报道的LG3上的QTL相同, 其它的均有较大差异:Caranta发现的位于Noir染色体上的QTL同up基因连锁, 以及发现的第3个QTL, 在Chaim等的研究中均没有发现(表1)。
马铃薯Y病毒:目前在辣椒上至少发现了7个单抗基因和1个QTL。1998年, Murphy等对抗马铃薯Y病毒基因pvr1进行了遗传定位〔7〕。1997年Caranta等将pvr2基因定位于P4连锁群上〔8〕。Tanksley进一步研究发现, 根据分子标记的连锁关系, pvr1、pvr2这两个基因是连锁的〔9, 10〕。同时有学者发现pvr3、pvr5没有连锁关系, 且同pvr1、pvr2也没有连锁关系〔7, 11, 12〕。1997年Caranta等在辣椒品种`CM334′ 上发现的pvr4基因抗PVY的所有小种, 为显性抗性〔12〕。1999年Caranta等开发了1个可以用于pvr4基因辅助选择的CAPS标记〔13〕。在辣椒中还发现了具有数量遗传性状控制的PVY抗性, 并进行了QTL分析, 发现共有9个QTL参与该性状控制, 其中有两个上位互作, 另有两个QTL位点与pvr2, pvr6位点重合〔8, 14〕(表1)。
番茄斑点萎蔫病毒:2000年Moury等利用分离组群分析法(BulkedSegregantAnalysis, BSA)和RAPD标记分析了含有153个单株的F2 群体, 开发了1个与番茄斑点萎蔫病毒(Tswv)紧密连锁的CAPS标记, 与Tswv的连锁距离为0.9 ±0.6 cM〔15〕(表1)。
1.1.2细菌性病害 疮痂病(Bacterialspot), 由野油菜黄单胞菌辣椒斑点病致病型(Xanthomonascampestrispv.vesicatoria)引起的。根据寄主的抗性反应, 病原菌划分了7个小种, 每个小种都有1个抗性基因对应。Bs1基因抗第0、2、5小种, Bs2基因抗0、3小种, Bs3基因抗0、1、4小种, 而小种6能克服以上3个基因。1999 年和2000 年Tai等和Pierre等利用SCAR标记和AFLP标记分别对Bs2和Bs3基因进行了定位, 其中Bs2 基因的标记完全与抗病基因共分离, 而Bs3基因定位于P2-Jaune染色体上〔16, 17〕(表1)。
1.1.3真菌性病害 辣椒疫病, 由辣椒疫霉菌(PhytophythoracapsiciLenioan)引起, 1918年由美国新墨西哥州首先报道, 中国在20世纪60年代报道, 目前在国内各省均有发生, 且危害程度逐年加重。抗疫病的遗传规律复杂, 在20世纪60 ~ 70年代针对部分材料的抗性归结为单隐性或双隐性或寡基因控制, 90年代进一步对部分抗性材料的研究表明, 辣椒对疫病的抗性受多个基因控制。1996年,Lefebvre等研究了辣椒材料Perennial对疫病的抗性规律, 确定了抗病基因在图谱上的QTL, 并认为参与抗性决定的位点有上位效应和加性效应〔18〕。2003年, Thabuis等将控制CM334、Van、Perennial, 3个抗病材料的抗疫病基因定位到染色体的18个位置上, 并认为存在上位作用〔19〕(表1)。
辣椒白粉病(PowderyMildew), 由鞑靼内丝白粉菌(Leveilluratauria)引起, 在20世纪70年代报道侵染辣椒等蔬菜作物, 近年来在辣椒上有加重的趋势。80年代, 在Capsicumannuum, Capsicum baccatum, Capsicumchinese上均发现了抗源材料〔20, 21〕。1989年Daubèze等发现了性材料`H3′ , 1995年又报道了其抗性是由寡基因控制〔22〕。2003 年, Lefebvre等将该抗性进行了QTL定位, 染色体P6、P9、P5、P10、P12上均有抗病位点, P2和P5有上位作用参与抗性决定〔23〕(表1)。
1.1.4 线虫 线虫(Nematodes)是辣椒的主要虫害之一, 在美国、地中海等地区均有发生, 中国主要发生在南方地区, 但随着保护地面积的增加, 北方有加重的趋势。根结线虫(Meloidogyne)是线虫中最重要的属, 有70多个种, 危害较严重的有南方根结线虫(M.incognita)、爪哇根结线虫(M.javanica)、花生根结线虫(M.arenaria)和北方根结线虫(M.hapla)等。目前, 在辣椒上已经发现并且命名的抗线虫基因至少有6个(n、Me1、Me2、Me3、Me4、Me5), 均为显性单基因控制, 其中Me3与Me4连锁, Me3对温度稳定〔24〕。2001 年Djian等利用AFLP技术和辣椒材料`YoloWonder’、`PM687′ 的DH群体完成了对Me3与Me4的分子遗传定位, 二者位于同一条染色体上, 相差9.9 cM, 并开发了1个与它紧密连锁的AFLP标记, 遗传距离为2.7 ±1.7 cM〔25〕, 见表1。

1.2 果实性状
辣椒果实相关性状多数是由多基因控制的数量性状, 最近对其遗传定位的研究比较多, 主要集中在果色、果实大小、果实形状、单果质量、辣味等方面。
2001年, Chaim等利用辣椒品种`Maor’ 和`Perennial’ 构成的F2 群体, 建成了一个177个分子标记、1 740 cM的遗传图谱, 进而对熟性、株高、单果质量、果实直径、果实长度、果形、果肉厚、可溶性固形物含量、胎座直径、胎座长度、种子质量、硬度、果实绿色变化、果实红色变化等多个性状进行了QTL定位〔26〕。表2显示了控制果实性状的QTL数目及其位置, 每个性状均有至少2 ~ 5个QTL参与了控制, 在染色体上某些部位较多的聚集了果实相关性状的QTL(P2、P3、P4、P8、P10), 这些区域在决定果实形状上无疑是非常重要的。
辣椒果色分为未熟期果色和成熟期果色。2003年Chaim等报道了对未熟期果实紫色的决定基因A、控制花药丝紫色的Fc基因和控制果形的主要的QTL进行分子遗传定位的结果。结果表明A基因与Fc基因位于染色体的同一位点或紧密连锁, 与TG63遗传距离为3 cM, 并与控制果宽和果形的QTL紧密连锁〔27〕。关于成熟期的果色, 红色对黄色是由单显性基因Y决定的。
1998年Lefebvre等将控制成熟期果色为红色/黄色的y基因定位到染色体Indigo上〔28〕, 并且认为该基因同Capsathin-capsorubin合成酶基因是相同的(表2)。1988年Tanksley等报道控制辣味有无的基因与CD35连锁〔29〕, 1995年Lefebvre等将其定位于连锁群7〔4〕。2001年Chaim等研究认为控制辣味有无的基因位于染色体2上, 最近的分子标记在10 cM以上〔6〕。2002年Blum等开发了与C基因紧密连锁的分子标记, 发现标记TG205与C基因共分离〔30〕,2003年又将辣椒素含量的QTL定位到染色体7上〔31〕(表2)。
果柄的方向受1对基因控制, 果柄向下对果柄向上是显性, 命名为up基因。1995年Lefebvre等将其定位于染色体P12, 与其最近的AFLP标记有5 cM的遗传距离〔4〕(表2)

1.3 雄性不育
目前报道的辣椒的雄性不育有两种, 一种是细胞核雄性不育(Genicmalesterility, GMS), 由单隐性基因控制;一种是胞质雄性不育(Cytoplasmicmalesterility, CMS)。胞质不育在多种植物中遗传稳定, 其遗传规律相对复杂, 它是由细胞核内的恢复基因和细胞质线粒体内的不育单基因共同作用的。关于辣椒细胞核内的恢复基因, 有单基因和双基因的争论, 但目前更多的学者倾向于是单主效基因控制、多微效基因修饰的作用结果。张宝玺等通过BSA法, 找到了与主效Rf基因连锁的RAPD标记, 这些标记对主效恢复基因辅助转育有一定帮助〔32〕;王立浩等利用DH分离群体, 通过分析育性的分离情况, 评价了CMS的遗传规律, 得出恢复性受主效基因和4个微效基因控制的结论, 并将主效基因定位到P6染色体上〔33〕。
2 辣椒的分子遗传图谱
2.1 辣椒分子遗传图谱的构建
辣椒分子遗传图谱的构建起步较早, 目前每年都有报道在已有图谱上添加新的标记, 或者创建新的遗传图谱。构建图谱的标记从同工酶发展到RFLP、RAPD、AFLP、SSR等。根据构建图谱材料双亲的血缘关系, 可以将图谱分为种内图谱和种间图谱。
2.1.1 种内图谱
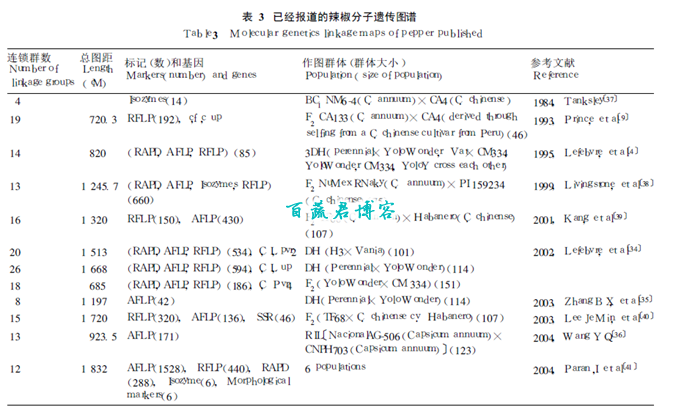
种内图谱采用的作图群体均来自于C.annuum。此类图谱的多态标记较少, 但对于育种更有意义。如表3所示, 法国农业科学院的Lefebvre等一直研究构建该类图谱, 1995年构建了1个包括了85个分子标记(RAPD、AFLP、RFLP)和控制抗烟草花叶病毒、果柄方向以及辣味基因的14个连锁群的分子遗传图谱, 总图距820 cM, 平均标记间距9.6 cM, 并将4个连锁群同染色体对应〔4〕。2002年, 又进一步丰富了该图谱, 在原有的基础上, 构建了3个分子标记遗传图谱, 分别将534、594和186个分子标记构建到20、26和18连锁群上, 添加了抗马铃薯Y病毒的基因, 图谱的总跨度分别达到了1 513、1 688、685 cM, 平均标记间距13、12.5、13.7 cM, 完成了6条连锁群与染色体的对应〔34〕。此外, 2003年张宝玺等构建了1个框架图谱, 8个连锁群, 图谱的精密度还有待进一步提高〔35〕;2004年, 亚蔬中心WangYongqing等利用1个含有123个单株的重组自交系(CIL)和AFLP标记技术, 构建了1个171个标记共13个连锁群, 总跨度923.5 cM的遗传图谱〔36〕。
2.1.2 种间图谱
种间图谱采用的作图群体来自于C.annuum和其它种, 如C.chinense。此类图谱的多态标记丰富, 更适合做高饱和图谱。康乃尔大学在构建辣椒的种间图谱方面做了大量工作。1984年, 康乃尔大学的Tanksley等构建的世界上第1个辣椒的分子遗传图谱即为种间图谱〔37〕;1993 年,Prince等利用CA133 (C.annuum)和CA4 (C.chinense)杂交和自交得到的包含了46个单株的F2群体, 构建了192个RFLP标记的连锁图谱, 共19个连锁群, 长720.3 cM, 其中还包括了控制花药丝颜色、果实辣味和果柄方向的3个基因〔9〕;1999年, Livingstone等利用1个含有75个单株的F2 群体, 构建了1 个13 个连锁群、460 个RFLP标记、7 个同工酶标记的遗传图谱, 总图距1 245.7cM〔38〕。韩国近年在图谱的建立和研究工作方面进展很快, 2001年和2003年, Kang等和Lee等分别利用RFLP、AFLP和SSR标记技术构建了两个分子遗传图谱, 总图距1 320 cM和1 720 cM〔39, 40〕。
2.1.3 图谱的整合 2004年, 以色列、美国康乃尔大学、法国农业科学院的Paran、Voort、Lefebvre等12名作者综合6个图谱的信息共同发表了1个相对完整的辣椒图谱, 包含了6个群体、2 262个分子标记, 总跨度1 832 cM, 平均标记间距0.8 cM, 大大增加了图谱的精密度〔41〕(表3)。
2.2 辣椒分子遗传图谱的比较
辣椒分子遗传图谱的比较研究开展得较早, 主要研究内容包括茄科蔬菜作物的同源性比较, 抗病基因位座比较, 亲缘关系比较等, 但利用图谱比较进行图谱构建和基因克隆的工作还不多。
2.2.1 辣椒与茄科其它蔬菜作物的同源关系 从早期的辣椒分子遗传图谱构建开始, 科学家们就利用图谱中的相同标记, 进行辣椒遗传图谱与番茄遗传图谱的比较, 后来扩大到整个茄科蔬菜作物。1993年, Prince等利用构建的辣椒分子遗传图谱与Tanksley等1992年构建的高密度的番茄RFLP图谱, 通过保守的RFLP标记进行了比较, 结果表明:辣椒的基因组同番茄的基因组具有大片段的同源, 同时高度重排〔9〕。进而1999年, Livingstone等利用其新建的辣椒分子遗传图谱与番茄、马铃薯和茄子的图谱通过相同的RFLP在图谱上的分布进行了12条染色体的深入比较〔39〕, 结果表明:辣椒上仅有4个区域没有在番茄上找到同源区域, 染色体2、6、7、10同番茄整条染色体同源, 部分辣椒染色体与2 ~ 3个番茄染色体同源, 染色体内存在不同程度的重排, 其中第6染色体并无大片段的重排, 而其它染色体与番茄的重排较多。因此得出结论:辣椒与番茄的基因含量大致相同;辣椒与番茄基因组由高度保守的片段组成;辣椒与番茄的图谱长度接近, 但辣椒的基因组约是番茄的3倍;一定数量的染色体重排决定了辣椒与番茄的差异。2002年, SamiDoganlar等构建了1个茄子(Solanummelongena)的分子遗传图谱〔42〕, 对比了Pillen发表的马铃薯(solanumtuberosum)遗传图谱、Tanksley发表的番茄(Lycopersiconesculentum)遗传图谱和Livingstone发表的辣椒遗传图谱, 得出结论:马铃薯与番茄的亲缘关系最近, 其次是与茄子, 最后是与辣椒。
2.2.2 茄科蔬菜作物抗病基因的位置关系 2000年, Grube等在茄科蔬菜作物间进行了抗病基因位点的比较, 利用Pillen发表的马铃薯遗传图谱、Tanksley发表的番茄遗传图谱和Livingstone发表的辣椒遗传图谱, 以及最近发表的抗病位点的定位, 再利用相同的分子标记进行位点比较, 绘制了图谱,将番茄、马铃薯、辣椒的抗病基因位点进行了很好的归纳, 同时表明多数的相同或类似病害的抗性基因在茄科蔬菜的基因组上分布的位置是保守的〔43〕。
3 问题与展望
作者认为在辣椒基因遗传定位及分子遗传图谱的研究方面还存在以下问题:1.目前辣椒分子遗传图谱的标记多为RFLP、AFLP等标记, 标记的种类比较单一, 一些稳定的、易于操作的标记还有待于开发, 连锁群和染色体的关系还没有一一对应。目前番茄的分子遗传图谱已经发展到1 668个分子标记, 并由RFLP、SSR、COS、EST、CAPS、KFG等多种分子标记组成。辣椒的基因组是番茄的3 ~ 5倍, 更需要高密度和多种标记的图谱。2.对新基因的定位不够。辣椒上很多基因的定位仅集中在几个作图群体, 种内图谱的抗病亲本材料主要有Perennial、CM334等。对于一些新材料含有的遗传机制不同的基因的定位并不多。3.对辣椒图谱的利用还不够, 通过比较对其它作物的图谱利用也不够。目前辣椒上的许多农艺性状的基因得到了分子定位, 抗病毒病、疫病、疮痂病等的性状, 果实大小、单果质量等产量相关性状等基因都得到了定位, 但目前国内还没有见到相关的应用研究报道。此外, 基因组比较的思路还没有很好的形成, 应用还不广泛。
传统遗传育种学同分子遗传学和细胞生物学的结合是作物遗传育种发展的必然趋势。国外利用分子标记的辅助选择在辣椒的抗疫病、抗PVY和品质育种上已有很多成功的范例, 而且从单基因分子标记辅助选择已经发展到QTL分子标记辅助选择。由于国内该领域研究起步较晚, 在分子标记辅助育种研究的广度和深度上同发达国家相比有明显距离。近年来, 越来越多的国内学者开始认识到分子标记辅助育种的重要性, “十五” 期间国家`863′ 项目对辣椒分子标记辅助育种也有了较大投入,经过努力, 我国已经在构建辣椒分子遗传图谱、辣椒雄性不育等重要性状的遗传定位方面取得了一些成绩。作者认为我国的辣椒分子标记和遗传图谱的研究还应该在以下方面努力:
1.丰富遗传图谱的标记数和种类。利用多种分子标记手段, 特别是基于PCR、易于操作的分子标记, 如:SSR、SNP、COS等标记, 利用高重组率的分离群体, 进一步丰富辣椒分子遗传图谱的标记。
2.在辣椒育种工作中积极实践分子标记辅助选择。充分利用已有辣椒图谱和重要性状的连锁标记, 进行辣椒的辅助选择。目前国内外报道了大量的与抗病性、丰产性、优良品质性状有关的分子标记, 有些与国内急需解决的问题有关, 例如抗疫病、抗CMV、雄性不育等, 这些标记应尽快应用到育种中。对于不大适用于大规模作为辅助育种的分子标记, 如:RFLP标记、AFLP标记等, 可以经过改造, 使之成为简单易行、相对稳定的PCR标记, 如:CAPS标记或SCAR标记等, 用于辅助育种。
3.开展针对新辣椒材料的图谱和分子标记工作。我国辣椒遗传资源丰富, 中期库拥有资源2 124份, 部分材料的抗病机制同国外已经报道的作图材料不同, 部分国内优秀的育种材料与国外品种有不同的农艺性状, 因此可以利用国内新的抗源材料或者优秀的育种材料构建分子遗传图谱, 此类图谱在育种工作上更有指导意义。4.在辣椒遗传育种研究中结合比较基因组的方法。通过比较基因组的方法, 可以利用番茄、马铃薯图谱上的分子标记丰富辣椒的遗传图谱;辣椒上某些基因的克隆可以借助图谱比较, 通过分析其它作物上的序列或图位, 得到更多基因的相关信息, 从而加快基因克隆的速度。
参考文献:
1 中华人民共和国农业部主编.中国农业统计资料.北京:中国农业出版社, 2003. 318 页
2 PetersonP.LinkageoffruitshapeandcolorgenesinCapsicum.Genetics, 1959, 44:407 ~ 419
3 BoukemaIW.ResistancetoTMVinCapsicumchacoensehunzisgovernedbyanalleleofthel-locus.CapsicumNewsl., 1982, 3:47 ~ 48
4 LefebvreV, PalloixA, CarantaC, PochardE.Constructionofanintraspecificintegratedlinkagemapofpepperusingmolecularmarkersand doubled-haploidprogenies.Genome, 1995, 38: 112 ~ 121
5 CarantaC, PalloixA, LefebvreV, DaubezeAM.QTLsforacomponentofpartialresistancetocucumbermosaicvirusinpepper:restriction ofvirusinstallationinhost-cells.Theor.Appl.Genet., 1997, 94: 431 ~ 438
6 ChaimAB, GrubeRC, LapidotM, JahnM, ParanI.IdentificationofquantitativetraitlociassociatedwithresistancetocucumbermosaicvirusinCapsicum annuum.Theor.Appl.Genet., 2001, 102: 1213 ~ 1220
7 MurphyJF, BlauthJR, LivingstoneKD, LackneyVK, JahnM K.Geneticmappingofthepvr1 locusinCapsicumspp.andevidencethat
distinctpotyvirusresistancelocicontrolresponsesthatdifferatthewholeplantandcellularlevel.Mol.Plant.Microb.Interact, 1998, 11(10):943 ~ 951
8 CarantaC, LefebvreV, PalloixA.Polygenicresistanceofpeppertopotyvirsusesconsistsofacombinationofisolatspecificandbroad-spectrum quantitativetraitloci.Mol.PlantMicrob.Interact, 1997, 10:872 ~ 878
9 PrinceJP, PochardE, TanksleySD.Constructionofamolecularlinkagemapofpepperandacomparisonofsyntenywithtomato.Genome, 1993, 36:404 ~ 417
10 GrubeRC, BlauthJR, ArnedoM S, CarantaAC, JahnM K.Identificationandcomparativemappingofadominantpotyvirusresistance geneclusterinCapsicum.Theor.Appl.Genet., 2000, 101:852 ~ 859
11 DogimontC, PalloixA, DaubèzeAM, MarchouxG, Gèbre-SelassieK, PochardE.Geneticanalysisofbroadspectrumresistancetopotyvirusesusingdoubledhaploidlinesofpepper(Capsicumannuum L.).Euphytica, 1996, 88:231 ~ 239
12 CarantaC, NemouchiG, DaubèzeA-M, PhalyT, PalloixA.ResistancetoPepMoVandPVY- 0 fromAvelararecontrolledbydistinctrecessivegenesandevidenceforindependencebetweenpvr3 andpvr5.Capsicum andEggplantNewsletter, 1999, 19:63 ~ 65
13 CarantaC, ThabuisA, PalloixA.DevelopmentofaCAPSmarkerforthePvr4 locus:Atoolforresistancetopyramidingpotyvirusresistance genesinpepper.Genome, 1999, 42:1111 ~ 1116
14 CarantaC, PlloixA.Bothcommonandspecificgeneticfactorsareinvolvedinpolygenicresistanceofpeppertoseveralpotyviuses.Theor. Appl.Genet., 1996, 92:15 ~ 20
15 MouryB, PfliegerS, BlattesA, LefebvreV, PalloixA.ACAPSmarkertoassistselectionoftomatospottedwiltvirus(TSWV)resistancein pepper.Genome, 2000, 43:137 ~ 142
16 TaiT, DahlbeckD, StallRE, PelemanJ, StaskawiczBJ.High-resolutiongeneticandphysicalmappingoftheregioncontainingtheBs2 resistancegeneofpepper.Theor.Appl.Genet., 1999, 99:1201 ~ 1206
17 PierreM, NoёlL, LahayeT, BallvoraA, VeuskensJ, GanalM, BonasU.High-resolutiongeneticmappingofthepepperresistancelocus Bs3 governingrecognitionoftheXanthomonascampestrispv.vesicatoraAvrBs3 protein.Theor.Appl.Genet., 2000, 101:255 ~ 263
18 LefebvreV, PalloixA.BothepistaticandadditiveeffectsofQTLsareinvolvedinpolygenicinducedresistancetodisease:acasestudythe interactionpepper-PhytophthoracapsiciLeonian.Theor.Appl.Genet., 1996, 93:503 ~ 511
19 ThabuisA, PalloixA, PfliegerS, DaubèzeA-M, CarantaC, efebvreV.ComparativemappingofPhytophthoraresistancelociinpepper germplasm:evidenceforconservedresistancelociacrosssolanaceaeandforalargegeneticdiversity.Theor.Appl.Genet., 2003, 106:1473 ~ 1485
20 UllassaBA, RawalRD, SohiHS, SingDP.Reactionofsweetpeppergenotypestoanthracnoseleafspotandpowderymildew.PlantDis.,1981, 65:600 ~ 601
21 DesphandeA, AnandN, PathakCS, SridharTS.NewsourcesofpowderymildewresistanceinCapsicum species.Capsicum Newslett,1985, 4:75 ~ 76
22 DaubèzeA-M, PochardE, PalloixA.ResistancetoLeveillulatauricainpepper(CapsicumannuumL.)isoligogenicallycontrolledandstableinMediterraneans.PlantBreeding, 1995, 114:327 ~ 332
23 LefebvreV, DaubèzeA-M, JRouppevanderVoort, PelemanJ, BardinM, PalloixA.QTLsforresistancetopowderymildewinpepperundernaturalandartificialinfections. Theor.Appli.Genet., 2003, 107:661 ~ 666
24 黄三文, 张宝玺, 郭家珍, 杨桂梅, 朱德蔚.辣(甜)椒根结线虫的危害、防治和抗病育种.园艺学报, 2000, 27 (增刊):515 ~ 521
HuangSW, ZhangBX, GuoJZ, YangGM, ZhuDW.Thedamage, controlandresistancebreedingofroot-knotnematodeinpepper.ActaHorticulturaeSinica, 2000, 27 (Suppl.): 515 ~ 521 (inChinese)
25 Djian-CaporalinoC, PijarowskiL, FazariA, SamsonM, GaveauL, OByrneC, LefebvreV, CarantaC, PalloixA, AbadP.High-resolution geneticmappingofthepepper(Capsicum annuumL.)resistancelociMe3 andMe4 conferringheat-stableresistancetoroot-knotnematodes (Meloidogynespp.).Theor.Appl.Genet., 2001, 103:592 ~ 600
26 Chaim AB, ParanI, GrubeR C, JahnM, WijkR V, PelemanJ.QTLmappingoffruit-relatedtraitsinpepper(Capsicum annuum). Theor.Appl.Genet., 2001, 102:1016 ~ 1028
27 ChaimAB, BorovskyY, JongW D, ParanI.LinkageoftheAlocusforthepresenceofanthocyaninandfs10.1 amajorfruit-shapeQTLin pepper.Theor.Appl.Genet., 2003, 106:889 ~ 894
28 LefebvreV, KuntaM, CamaraB, PalloixA.Thecapsanthin-capsorubinsynthasegene:acandidategenefortheylocuscontrollingthered fruitcolourinpepper.PlantMol.Biol., 1998, 36:785 ~ 789
29 TanksleySD, BernatzkyR, LapitanNL, PrinceJP.Conservationofgenerepertoirebutnotgeneorderinpepperandtomato.Pro.Natl.AcadSci.U.S.A., 1988, 85:6419 ~ 6423
30 BlumE, LiuK, MazourekM, YooEY, JahnM, ParanI.MolecularmappingoftheClocusforpresenceofpungencyinCapsicum.Genome,2002, 45:702 ~ 705
31 BlumE, MazourekM, OConnellM, CurryJ, ThorupT, LiuK, JahnM, aranI.Molecularmappingofcapsaicinoidbiosynthesisgenesand quantitativetraitlocianalysusforcapsaicinoidcontentinCapsicum.Theor.Appli.Genet., 2003, 108:79 ~ 86
32 ZhangBX, HuangSW, YangGM, GuoJZ.TwoRAPDmarkerslinkedtoamajorfertilityrestorergeneinpepper.Euphytica, 2000, 113:155 ~ 161
33 WangLH, ZhangBX, LefebvreV, HuangSW, DaubèzeAM, PalloixA.QTLanalysisoffertilityrestorationincytoplasmicmalestrile pepper.Theor.Appli.Gene., 2004, 109:1058 ~ 1063
34 LefebvreV, PfiegerS, ThabuisA, CarantaC, BlanttesA, ChauvetJ-C, DaubèzeA-M, alloixA.Towordsthesaturationofthepepperlinkagemapbyalignmentofthreeintraspecificmapsincludingknown-functiongenes.Genome, 2002, 45:839 ~ 854
35 张宝玺, 王立浩, 黄三文, 郭家珍, 杨桂梅, 堵玫珍.辣椒分子遗传图谱的构建和胞质不育恢复性的QTL定位.中国农业科学,
2003, 36 (7):818 ~ 822 ZhangBX, WangLH, HuangSW, GuoJZ, YangGM, DuMZ.ConstructionofamolecularlinkagemapandQTLanalysisoffertilityrestorerofCapsicum annuum L.ScientiaAgriculturaSinica, 2003, 36 (7): 818 ~ 822 (inChinese)
36 WangYQ.Constructionofamoleudarlinkagemapofpepper(Capsicum sp.) usingAFLPmarkersandRILpopulation2004, http: //www.arc-acrde.orglhtml-files/moleaderhtml
37 TanksleySD.Linkagerelationshipsandchromosomallocationsofenzyme-codinggenesinpepper, Capsicumannuum.Chromosoma, 1984,89:352 ~ 360
38 LivingstoneKD, LackneyVK, BlauthJR, WijkRV, JahnM K.GenomemappinginCapsicumandtheevolutionofgenomestructurein solanaceae.Genetics, 1999, 152:1183 ~ 1202
39 KangBC, NahmSH, HuhJH, YooH S, YuJW, LeeM H, KimBD.Aninterspecific(Capsicumannuum×C.chinense)F2 linkage mapinpepperusingRFLPandAFLPmarkers.Theor.Appl.Genet., 2001, 102:531 ~ 539
40 LeeJM, KimBD.CombinedgenomemappingofRFLP-AFLP-SSRinpepper.GenomicsandInformatics, 2003, 1 (2):108 ~ 112
41 ParanI, JRvanderVoort, LefebvreV, JahnM, LandryL, M vanSchriek, TanyolacB, CarantaC, ChaimAB, LivingstoneK, PalloixA,
PelemanJ.Anintegratedgeneticlinkagemapofpepper(Capsicumspp.).MolecularBreeding, 2002, 13:251 ~ 261 42 DoganlarS, FraryA, DaunayM C, LesterRN, TanksleySD.Acomparativegeneticlinkagemapofeggplant(Solanummelongena)andits implicationsforgenomeevolutionintheSolanaceae.Genetics, 2004, 161:1697 ~ 1711
43 GrubeRC, RadwanskiER, JahnM.ComparativegeneticsofdiseaseresistancewithintheSolanceae.Genetics, 2000, 155:873 ~ 887
转载请注明:百蔬君 » 美文分享:辣椒基因遗传定位及分子遗传图谱的研究进展



